近日,猪禽种业全国重点实验室、华南农业大学动物科学学院张哲教授团队在国际顶级学术期刊《自然-通讯》(Nature Communications,IF5=17.2)在线发表题为“OmiGA for ultra-efficient molecular quantitative trait loci mapping”的研究论文。团队成功研发出基于线性混合模型的高性能分子数量性状基因座(分子QTL)定位方法OmiGA(官网:https://omiga.bio/)。该方法针对家养动物复杂亲缘关系群体的分子QTL定位难题实现突破,在运算效率、因果变异鉴定准确性上表现优异,全方位超越主流分析方法。

分子QTL定位是解析生物复杂性状与疾病遗传调控机制的核心手段。然而,现有工具多依赖简单线性模型,难以适配复杂亲缘关系分析场景,常面临假阳性偏高、统计效力不足的瓶颈。针对这一难题,团队搭建了稳健的方法体系并开发了拥有自主知识产权的OmiGA工具。OmiGA功能体系完备,当前包含两大核心模块:一是分子QTL定位模块,支持顺式(cis)、反式(trans)及环境依赖效应分析,可精准解析加性、非加性遗传效应;二是高通量分子表型遗传力估计模块,能实现加性和非加性效应下,顺式、反式及全基因组层面的遗传力精准测算。
OmiGA兼具了超高速和高精准的核心优势。OmiGA在分子性状遗传力估计中的速度相较于GCTA、LDAK等主流工具最高提升1600余倍。OmiGA鉴定的顺式表达数量性状基因座(cis-eQTL)因果概率显著高于主流的tensorQTL;以瘦肉率性状与十二指肠TRIM52基因为例,OmiGA结果的共定位概率高达0.84,而tensorQTL仅为0.05,与模拟研究结果共同证明了OmiGA能更精准地鉴定出贴近真实的因果遗传变异。
OmiGA突破了非加性分子QTL分析的盲区。现有工具大多仅能检测加性遗传效应,而OmiGA创新性地将显性模型全流程纳入分析框架。在人源淋巴母细胞系(MAGE LCL)数据中,OmiGA成功捕捉到常染色体显性多囊肾病相关基因PKD1的显性变异rs238683(P=3.08×10-8),而加性模型在此完全失效。这一突破为解析非加性遗传调控效应机制提供了关键技术。
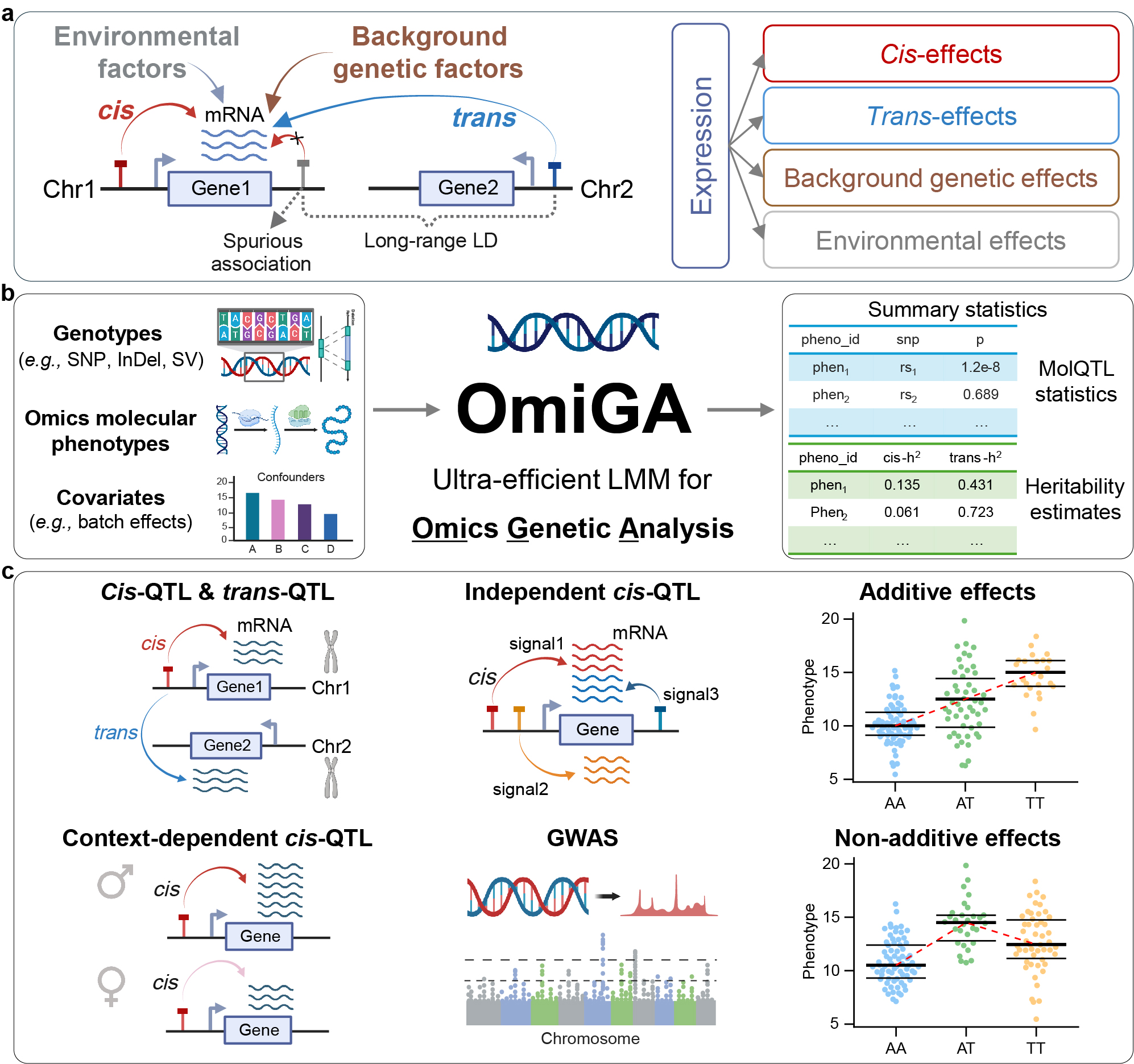
OmiGA的问世为多组学大队列数据的超快速遗传分析提供了核心方法支撑,将有力推动国际大科学计划FarmGTEx及农业生物复杂性状的遗传解析工作,为畜禽种业分子育种注入了全新的技术动力。
动物科学学院首聘副教授滕金言、博士生张文劲为论文共同第一作者,张哲教授、滕金言副教授与丹麦奥胡斯大学房灵昭教授为共同通讯作者。本研究获农业科技重大专项(2022)、中国农业产业技术体系(CARS-35)资助,国家超算无锡中心为研究提供了计算支持。
相关论文链接:https://doi.org/10.1038/s41467-026-68978-0
文图/动物科学学院



