近日,资源环境学院仇荣亮教授团队在Nature杂志子刊Nature Communications(中科院1区,5年影响因子17.2)在线发表题为“Potentiators empower synthetic microbiomes as silent guardians against co-contamination”的研究论文。该研究围绕复杂有机污染情景下合成微生物组的理性构建与功能强化,提出了一种全新的菌群设计范式,为高效、稳定的环境生物修复提供了重要理论依据与技术支撑。

环境中多种有机污染物共存已成为当前生态修复领域面临的突出挑战。相比单一菌株,微生物群落在功能冗余、代谢互补与协同降解方面具有显著优势,但如何在复杂胁迫条件下构建兼具高效性与稳定性的合成菌群,仍缺乏系统性的设计原则。针对这一问题,仇荣亮教授团队基于自主研发的基因组尺度代谢模型工具 SuperCC,系统模拟了由多株关键功能菌组成的合成菌群在单一污染物与复合污染条件下的代谢行为,深入解析了污染物—菌株—代谢网络之间的互作机制。
研究通过整合多组学数据与代谢建模分析,揭示了复合污染胁迫下菌群协同作用的关键驱动机制。结果表明,具有广谱资源利用能力的菌株通过分泌特异性代谢产物,在群落中发挥“增效”作用,是促进菌群协同降解的重要物质基础,不仅仅提高整个群落的生物量,还增强了菌群的降解功能;同时,本土关键菌株在维持群落结构稳定性和功能持续性方面具有不可替代的核心作用。在此基础上,研究创新性提出了“降解菌—辅助菌—增效菌”协同构建的合成菌群设计范式(DHP-Com),显著提升了合成微生物组在多类型复合污染场景下的修复效率与生态适应性。
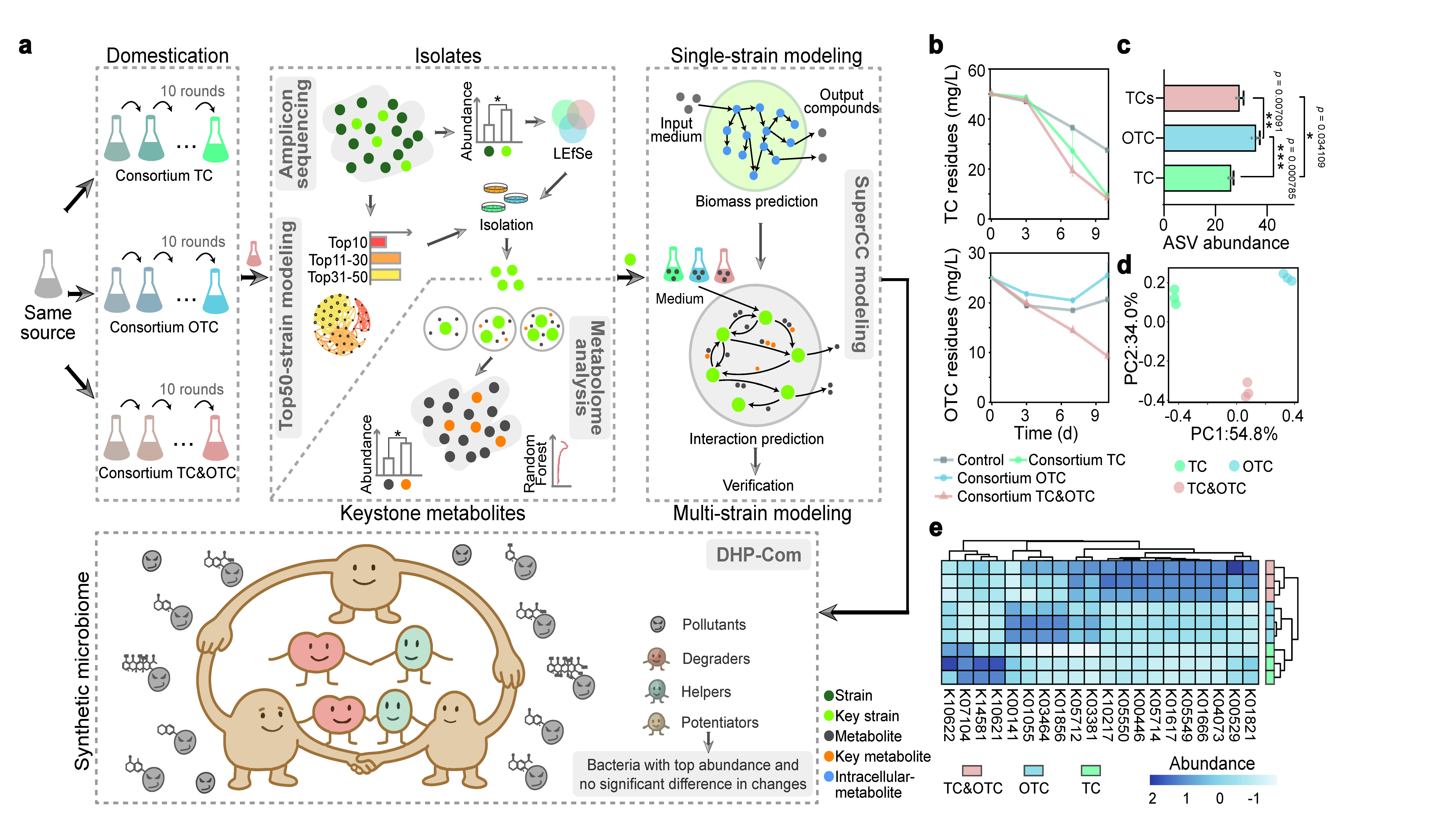
图1 复合抗生素污染下的实验设计与微生物菌群响应
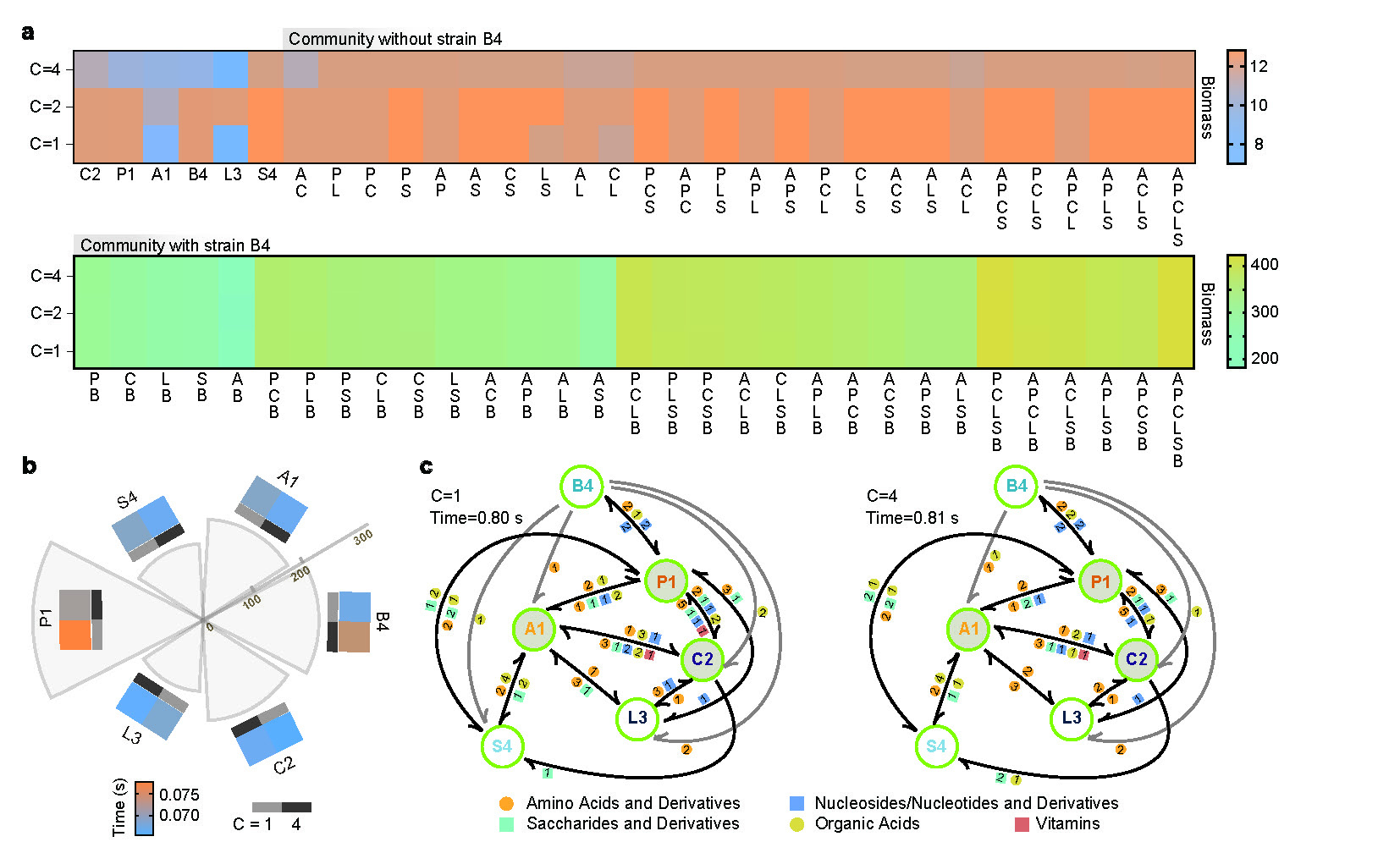
图2 合成微生物组的多环境条件模拟与互养作用分析
该研究推动了基因组尺度代谢模型在复杂环境污染修复中的应用,从经验式菌群组合迈向模型驱动的理性设计,为构建多功能、高稳定性的合成微生物组提供了系统化思路,也为应对现实环境中普遍存在的复合有机污染问题提供了新的解决路径。
资源环境学院副教授阮哲璞和博士生谭家霖为该论文的共同第一作者,仇荣亮教授为通讯作者。第一通讯单位为华南农业大学、岭南现代农业科学与技术广东省实验室、广东省农业与农村污染防治及环境安全重点实验室。研究得到国家重点研发计划(2023YFC3905800、2023YFC3707600)、国家自然科学基金(42307006)、广东省重点领域研发计划(2023B0202020001)及广东省科技计划项目(2021B1212040008 )资助。
论文相关链接:https://www.nature.com/articles/s41467-025-67953-5#citeas
文图/资源环境学院



