近日,亚热带农业资源保护与利用国家重点实验室夏瑞团队在Genome Biology(影响因子10.1)上在线发表了题为“SynGAP: a synteny-based toolkit for gene structure annotation polishing”的研究论文。该研究开发了一种基于基因共线性进行物种基因组基因结构注释矫正的工具SynGAP(Synteny-based Gene structure Annotation Polisher)。该工具基于近缘物种基因共线性,去鉴定并矫正原始基因结构注释中的潜在错漏,实现基因结构注释的优化。

演化过程中,在具有共同祖先的近缘物种之间,染色体上同源基因存在保守排列的现象,被称为基因共线性(gene synteny)。近缘物种的基因共线性区块中,部分基因丢失了与其对应的共线性基因,进而在区块内形成共线性对的间隔(gap,图1a)。共线性基因的缺失,可能由基因组序列的变化引起的,同时还有可能是错误注释或缺失的基因模型(mis-annotated or absent gene models,MAGs)导致的。基于后一种可能性,作者们通过两物种的共线性分析,检测出共线性区块中共线性对的空缺位置(gap区域)。随后进行双向的同源比对以实现对gap内潜在注释错漏的初步鉴定与矫正。再通过去冗余、可靠性指标(R value)计算筛选、参考注释质量分级等步骤对初步矫正结果进行质控,最终获得两物种的高质量矫正注释,并且实现对gap的填补。通过多个植物、动物物种组合的测试与统计,明确SynGAP dual可以对被测试基因组GSA进行优化——增加优质新基因注释以及共线性基因对,同时提高了BUSCO完整度。
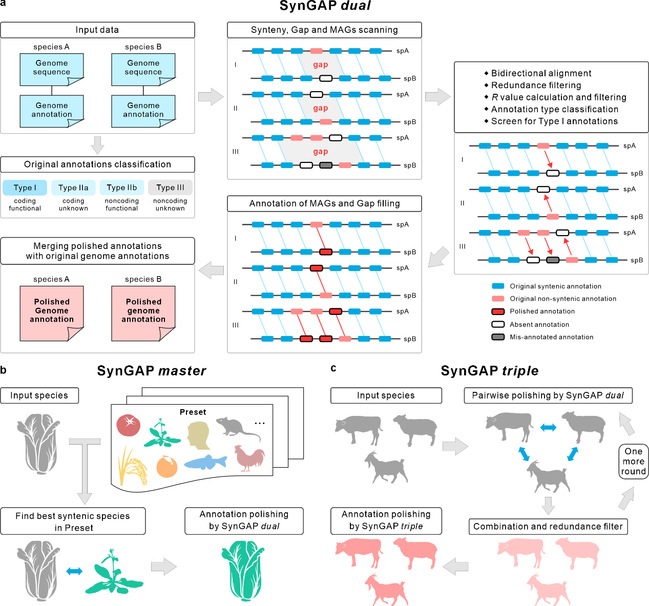
SynGAP基因结构注释矫正的设计逻辑与流程
除了基因结构注释矫正功能模块外,SynGAP还包含了一套基因物种比较转录组分析流程(包含genepair和evi模块)。通过该流程可实现近缘物种间的准确基因配对,并结合转录组数据完成跨物种时序性转录组分析,高效地筛选鉴定候选关键差异表达基因。其中设计了EVI这一基因差异表达指标,可同时体现物种间对应基因的表达水平差异、表达量倍数差异以及表达模式变化差异。基因对的EVI值越高,两个同源基因的差异表达就越显著。经测试,EVI可以作为鉴定控制特定性状或发育过程(如花色素苷合成、辣椒素合成、内果皮木质化和大脑体积增大)的候选关键基因的有效指标。
本论文以华南农业大学为第一完成单位,园艺学院夏瑞教授、陈程杰博士为共同通讯作者。博士研究生吴锋琦为该论文第一作者。博士研究生麦迎晓参与了该论文的软件功能设计。该研究得到“十四五”广东省农业科技创新十大主攻方向“揭榜挂帅”项目、国家自然科学基金、广东省重点研发项目等资助。
文图/亚热带国家重点实验室

